Biogeographie, genetische und funktionelle Diversität von Pflanzenwachstum-fördernden methylotrophen Bakterien
Methylotrophe Bakterien sind Mikroorganismen, die als Kohlenstoff- und Energiequelle Methan und Methanol nutzen können. Sie kommen unter anderem auf Blättern von Pflanzen vor und leisten dort einen wichtigen Beitrag im Methanolkreislauf, indem sie das von den Pflanzen produzierte Methanol umwandeln. Zusätzlich können methylotrophe Bakterien für die Pflanze essentielle und wachstumsfördernde Substanzen wie z.B. Auxine, Cytokinine und Vitamin B12 produzieren.
Über die Diversität und die Verbreitung von methylotrophen Bakterien ist wenig bekannt. In diesem Projekt soll daher die genetische Diversität, Biogeographie und funktionelle Rolle der auf Pflanzen vorkommenden methylotrophen Bakterien im Zusammenhang mit den besiedelten Pflanzenarten, dem Landnutzungstyp und der geographischen Lage untersucht werden. Da die Untergruppe der pink-pigmentierten fakultativ methylotrophen Bakterien (PPFM) mit bis zu 79% der gesamten Heterotrophen in der Phyllosphere von Pflanzen die größte Bedeutung hat, wird auf diese Gruppe das Hauptaugenmerk gelegt.
1. Die Diversität methylotropher Bakterien ändert sich in Abhängigkeit von der Landnutzungsintensität, der besiedelten Pflanzenarten und der geographischen Distanz
2. Die Produktion von für die Pflanze wachstumsfördernder Substanzen durch methylotrophe Bakterien ist abhängig von der Zusammensetzung der Bakteriengemeinschaft und von der Landnutzungsintensität
3. Die genetische Variabilität innerhalb einer Art ist bei methylotrophen Bakterien abhängig von der Landnutzung und / oder der geographischen Distanz
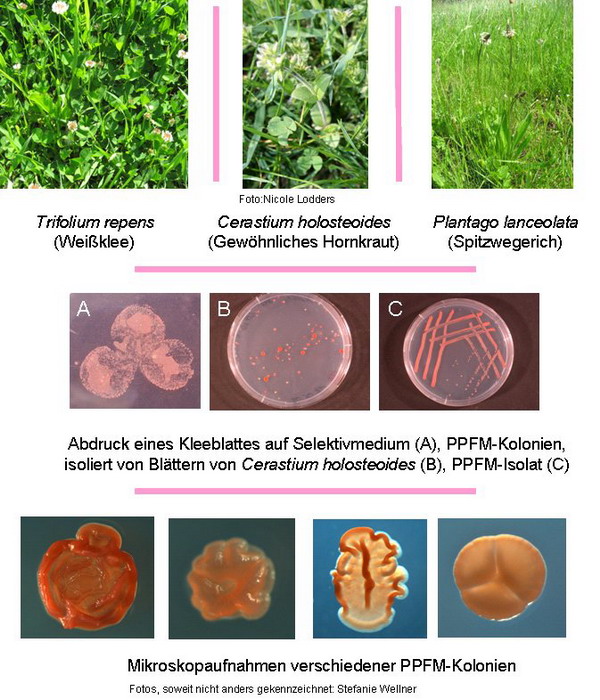
Es wurden Blattproben von zuvor ausgewählten Pflanzen, Trifolium repens (Weißklee), Cerastium holosteoides (Gewöhnliches Hornkraut) und Plantago lanceolata (Spitzwegerich), von Flächen unterschiedlicher Landnutzungstypen gesammelt. Zur Bestimmung der Konzentration der PPFM pro Gramm Blatt wurde eine Kultivierung auf einem mit Methanol angereicherten Selektivmedium durchgeführt. Von repräsentativen Isolaten wurden die 16S rRNA-Gene sequenziert und die erhaltene Sequenzinformation für phylogenetische Analysen herangezogen. Zur Untersuchung der Diversität innerhalb ausgewählter methylotropher Arten wurde das mxaF-Gen sequenziert und analysiert. Das mxaF -Gen kodiert für die ?-Untereinheit der Methanoldehydrogenase, die in allen Methylotrophen präsent ist. Weiterhin sollen Diversitätsänderungen der verschiedenen Umweltproben mit sogenannten fingerprinting Methoden (DGGE) untersucht werden.
Erste Ergebnisse sind:
- Landnutzungstypen und Pflanzenarten haben einen signifikanten Einfluss aufdie Konzentration von methylotrophen Bakterien auf Blattoberflächen.
- Fünf phylogenetisch verschiedene Methylobacterium Gruppen einschl. drei neuer Arten wurden gefunden.
- Die Artenzusammensetzung der methylotrophen Bakterien ist nicht durch die Landnutzung beeinflusst.