Prokaryotische Diversitätsveränderungen und ihre funktionelle Wechselbeziehung zur Landnutzung
Die Acidobacteria bilden ein weit verbreitetes und abundantes aber wenig untersuchtes Phylum der Bakterien. Anhand von molekularen Studien der 16S rRNA-Gene konnten Acidobacteria in verschiedenen Ökosystemen wie z. B. in Böden und Sedimenten, in heißen Quellen, im Torfmoor, im Plankton des tiefen Mittelmeeres, in sauren Seen von Abraumhalden und in Höhlen nachgewiesen werden.
Acidobacteria können bis 50 bis zu 80% der Bodenbakterien ausmachen. Es wird angenommen, dass die phylogenetische Diversität der Acidobacteria und ihre Abundanz im Boden ähnlich groß sind wie die des Phylums Proteobacteria. Diese phylogenetische Diversität und die weite Verbreitung und Abundanz vor allem in Bodenhabitaten deuten auf eine wichtige Rolle bei biogeochemischen Prozessen und eine hohe metabolische Vielseitigkeit dieser Bakterien hin. Über die funktionelle Rolle der Acidobacteria und den Zusammenhang zur Landnutzung ist jedoch noch fast nichts bekannt.
1. Acidobacteria sind eine dominante Gruppe von Bodenbakterien und relevant für die Ökosystemfunktionen in Böden.
2. Die Diversität und die Aktivität der Acidobacteria korrelieren mit der Pflanzendiversität.
3. Die Landnutzung ist eine Einflußgröße der funktionellen Diversität der Acidobacteria.
In dem vorliegenden Projekt soll über einen Landnutzungsgradienten als Hauptvariable die funktionelle Relevanz von Diversitätsänderungen untersucht werden. Zielsetzungen sind die Erfassung der Zusammensetzung, der physiologischen Schlüsselfunktionen und die Identifizierung wichtiger funktioneller Gruppen der Acidobacteria.
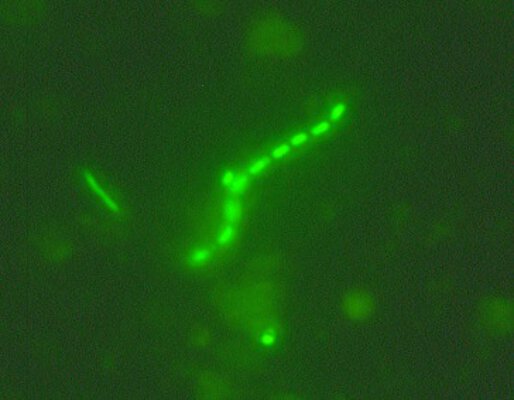
Diversitätsänderungen und physiologisch aktive Phylotypen werden mit sogenannten fingerprinting Methoden, DGGE und T-RFLP, untersucht. Um dominierende Populationen zu identifizieren, werden 16S rRNA-Klonbibliotheken angelegt und entsprechende Klone sequenziert. Zur Erfassung der physiologischen Eigenschaften werden existierende Metagenom-Bibliotheken genutzt und repräsentative Vertreter mit neuartigen Kultivierungsmethoden isoliert. Durch die Zugabe verschiedener 13C-Kohlenstoffquellen und dem Verfolgen des Einbaus dieser Substrate mittels stable isotope probing der RNA soll die funktionelle Rolle einiger wichtiger Phylotypen aufgeklärt werden. Ausserdem untersuchen wir mittels Zellzählungen, DGGE und T-RFLP-Analyse die jahreszeitliche Variation der Acidobacteria-Gemeinschaften und beproben im April, Juni, August und Oktober 2009 alle 57 VIP-Plots.
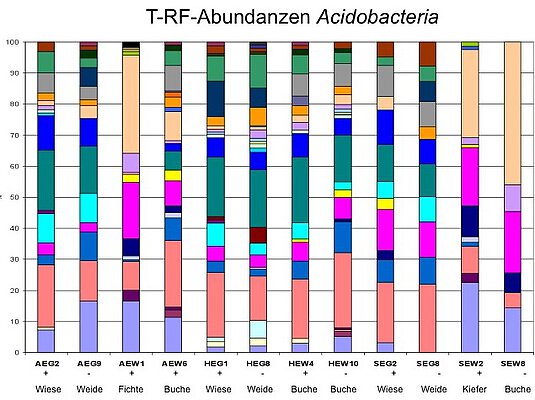
Zellzählungen zeigten bisher, dass im Exploratorium Schwäbische Alb die meisten Zellen je g Boden auftreten, gefolgt vom Hainich-Dün und der Schorfheide-Chorin. Die Clusteranalysen der durch DGGE und T-RFLP-Analysen aufgenommenen acidobakteriellen Gemeinschaften zeigten, dass Bodenproben eines Exploratoriums generell zusammen clustern, aber durch den Typ der Landnutzung (Wald/Grünland) getrennt werden. Die ungleiche Verteilung und Abundanz der Acidobacteria in Wald und Grünland führt so zu getrennten Clustern. Besonders die Waldböden der Schorfheide unterscheiden sich signifikant von allen anderen Böden und bilden ein eigenes Cluster. Das Vorkommen der Acidobacteria ist stark pH-abhängig; demzufolge resultieren diese Ergebnisse wohl aus unterschiedlichen pH-Werten in Wald (~ pH 5.0) und Grünland (~ pH 6.5), wobei der Waldboden im Exploratorium Schorfheide-Chorin mit Abstand die niedrigsten pH-Werte (~ pH 3.5) zeigte.