BELongDead – Multitrophische funktionale Diversität im Totholz
Totholz ist eine wichtige Kohlen- und Nährstoffquelle in Waldökosystem und wird kontinuierlich durch Makro- und Mikroorganismen abgebaut. Hierdurch werden Waldökosysteme maßgeblich strukturiert und viele Ökosystemprozesse beeinflusst. Obwohl in den letzten Jahren zahlreiche Studien zur Diversität einzelner Artengruppen in Totholz erschienen sind, fehlen uns immer noch umfassende Kenntnisse, wie holzbewohnende Organismen im zeitlichen Kontext organsiert, strukturiert und miteinander vernetzt sind und dadurch den Abbau des Totholzes entscheidend beeinflussen.
Unser Projekt ist Teil des BELongDead-Konsortiums. Wir fokussieren uns auf alle relevanten und zugrunde liegenden molekularen und biochemischen Prozesse des organismisch-beeinflussten Totholzabbaus im Zusammenhang mit Waldbewirtschaftungsintensitäten und räumlich-geographischer Skalierung.
Insbesondere soll untersucht werden:
- wie Arten Totholzstämme besiedeln und sich etablieren,
- wie Arten unterschiedlicher taxonomischer Gruppen im Verlauf der Sukzession miteinander interagieren,
- wie sich der Einfluss der Baum- und somit Holzeigenschaften und der Umgebung/Umwelt über die Sukzession verändert und
- ob sich kausale Zusammenhänge zwischen Diversität und Abbau besser verstehen lassen, wenn man möglichst viele Organismengruppen betrachtet.
Wir nutzen klassische, sowie modernste molekularbiologische Methoden und ´Genomics´-Ansätze, um möglichst viele Aspekte biologischer Diversität zu erfassen (Abb. 1). Diese Daten werden schließlich mit Informationen über Holzchemie, Enzymaktivitäten und dem Abbau des Holzes im Allgemeinen verknüpft. Wir erfassen folgende Daten:
- DNA-basierte Diversität von Pilzen, Bakterien, Archeen und Nematoden
- Diversität von Pilzfruchtkörpern, Flechten und Moosen (Abb. 2)
- Holzcharakteristika wie pH-Wert, C/N-Verhältnis, Ligningehalt
- Holzabbau-relevante Enzymaktivitäten (z.B. Peroxidasen, Laccasen)
Diese Daten werden an zwei verschiedenen Experimenten erhoben:
a) BELongDead
Nach den Probennahmen 2012, 2015 und 2017 wird nun 2020 unsere Datenreihe durch einen vierten Zeitpunkt erweitert, um die Prozesse des Holzabbaus von der initialen bis zur teils weit fortgeschrittenen Zersetzungsphase bewerten zu können.
b) BESterile
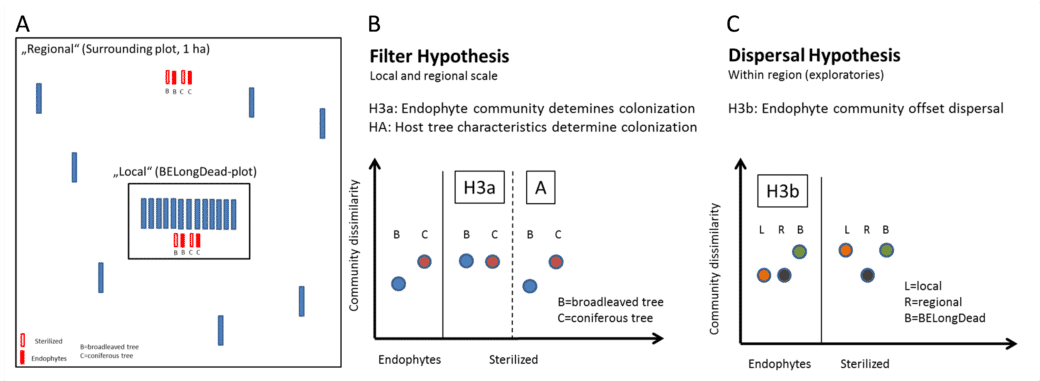
In einem neuen Zusatzexperiment werden Gamma-sterilisierte und unbehandelte Totholzstämme der beiden Hauptbaumarten mitteleuropäischer Wälder Fagus sylvatica und Picea abies neben den vorhandenen BELongDead-Stämmen sowie in einem genau definierten Abstand ausgelegt (Abb. 3A). So wollen wir zwei grundlegende Forschungsfragen beantworten:
- Zeigt ein Vergleich zwischen den nicht sterilisierten Stämmen (gekennzeichnet durch die Endophytengemeinschaft) und den sterilisierten Kontrollen einen Unterschied in der kolonisierenden Diversität und schlussfolgernd in der Holzabbaurate? Werden die Unterschiede zwischen verschiedenen Baumarten hauptsächlich durch die Endophytengemeinschaften (Abb. 3B, Hypothese H3a) oder alternativ durch die Eigenschaften des Wirtsbaums (z.B. physikochemische Eigenschaften des Holzes, Abb. 3B, Hypothese H3A) verursacht?
- Wir erwarten, dass die Kolonisierung vom Spenderpool abhängt und somit Unterschiede in den Besiedlungsmustern zwischen den Stämmen direkt an BELongDead (stärker am lokalen Artenpool) und jenen in weiter Entfernung auftreten (stärker am regionalen Artenpool). Wenn die endophytische Gemeinschaft die Kolonisierung bestimmt, werden wir eine ähnlichere Artengemeinschaft zwischen allen Stämmen beobachten (Abb. 3C, Hypothese H3b).