Die Funktion der Hyphosphäre bei der Kohlenstoff- und Nährstoffverteilung zwischen Pflanzen und Mikroorganismen in Grünlandböden mit unterschiedlicher Landnutzungsintensität
Eine Verringerung der Landnutzungsintensität kann Interaktionen zwischen Pflanzen und Symbionten begünstigen und so die Strategien zur Nährstoffaufnahme von Pflanzen und Mikroben auf verschiedenen Skalen verändern. Auf der Skala der Rhizosphäre, zum Beispiel einem schmalen Bereich des Bodens, der direkt von Wurzelsekreten und assoziierten Bodenmikroorganismen beeinflusst wird, ist die Wurzel-Boden-Grenzfläche durch Gradienten von Nährstoffen, Protonen, Kohlenstoffressourcen und lebenden Mikroorganismen gekennzeichnet. Die Hyphosphäre ist definiert als ein Bereich, in dem symbiotische und nichtsymbiotische Pilze in der Bodenumgebung vorhanden sind; sie ist gekennzeichnet durch intensive Pilz-Bakterien-Böden. Diese unterschiedlichen Zonen lassen sich experimentell oft nur schwer voneinander trennen.
Ziel unserer Studie ist es, zu erforschen, wie Pilz-Bakterien-Interaktionen in der Hyphosphäre zur Pflanzenernährung beitragen und wie sich die Kohlenstoffversorgung durch die Pflanzen über die symbiotischen Interaktionen mit Pilzen auf Bakterien auswirkt.
In den nächsten drei Jahren werden wir die folgenden Hypothesen testen:
- Sowohl symbiotische (Mykorrhizapilze) als auch freilebende saprotrophe Pilze dominieren die anfängliche mikrobielle Kohlenstoffaufnahme der Pflanze durch schnelle Verarbeitung und Kanalisierung von Rhizodeposite in die Hyphosphäre. Folglich kann die Hyphosphäre als “Kohlenstoffbrücke” zwischen Pflanzen und Bakterien fungieren; Bakterien profitieren als sekundäre Konsumenten.
- Pflanzen sind auf den Nährstofftransport über die Hyphosphäre stärker in Grünlandböden mit niedriger als mit hoher Landnutzungsintensität (LUI) angewiesen.
- Eine experimentelle Extensivierung der Grünlandnutzung wird die Abundanz und Funktion der Bodenmikroorganismen verändern, entweder durch Verringerung des Nährstoffangebots (verringerte Düngung, direkte Reaktion der Bodenmikroorganismen) oder durch Verringerung der Mähintensität (Veränderung des Pflanzeninputs in den Boden, indirekte Reaktion der Bodenmikroorganismen).
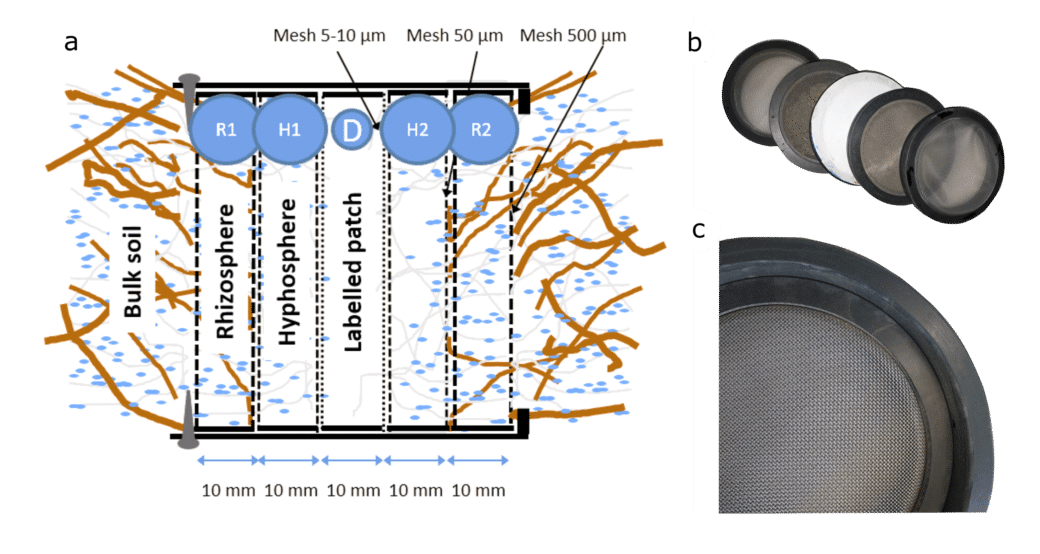
Um unsere Hypothesen zu überprüfen, brauchen wir eine vollständige Trennung zwischen Rhizosphäre und Hyphosphäre. Wir werden hierzu neu entwickelte HYPHO-Boxen verwenden, die die Hyphosphäre experimentell von der Rhizosphäre und dem umgebenden Boden trennen (Abb. 1).
Experiment 1 und 2: Kohlenstofffluss von den Pflanzen in die Hyphosphäre und Nährstofffluss von der Hyphosphäre zu den Pflanzen
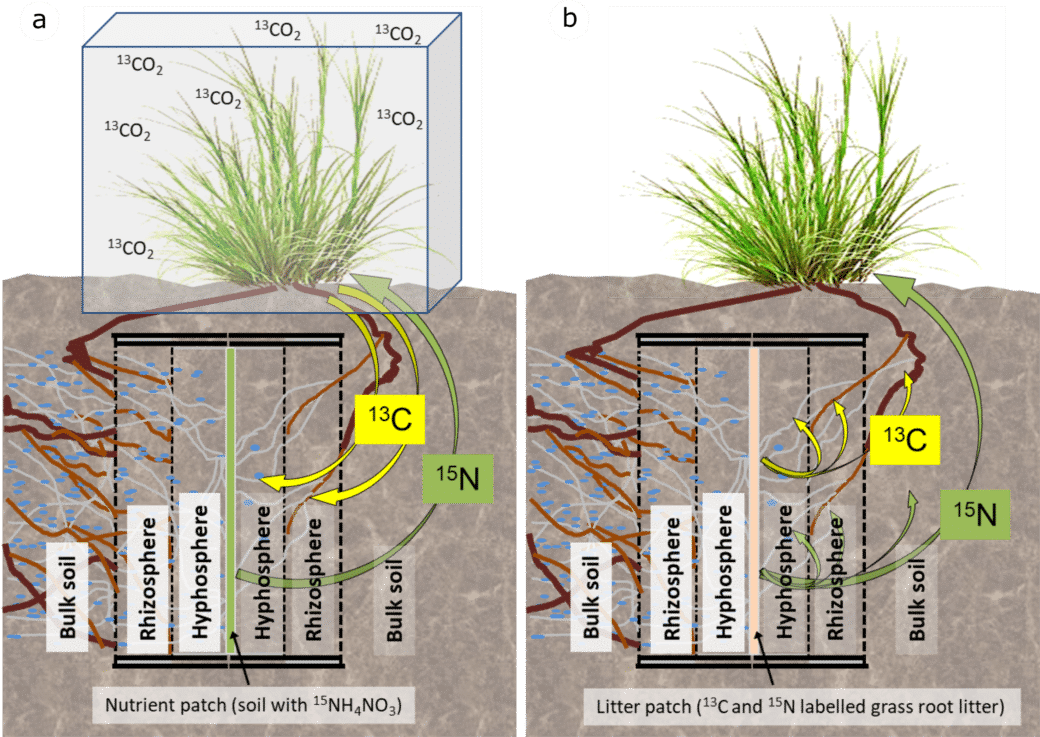
Ziel dieser Experimente ist es zu verstehen, (i) welche Pilze in Grünlandökosystemen am meisten vom pflanzlichen Kohlenstoff profitieren und welche Pilze für den “Langstrecken”-Transport (>15 mm) von Kohlenstoff verantwortlich sind und damit Ressourcen für Bakterien als sekundäre Konsumenten bereitstellen (Abb. 2a); und (ii) welche Pilze für den Transport von 13C und 15N von der Detritusphäre durch die Hyphosphäre in die Rhizosphäre verantwortlich sind (Abb. 2b). Wir werden einen 13CO2-Pulsmarkierungsansatz verwenden, um den Kohlenstofffluss von pflanzenfixiertem C in die Pilze und ihre assoziierten Bakterien zu quantifizieren, und eine 13C- und 15N-Markierung im Detritusphärenkompartiment wird es uns ermöglichen, den langfristigen 13C- und 15N-Einbau in die mikrobielle Biomasse sowie deren Übertragung auf die pflanzliche Biomasse zu quantifizieren.
Experiment 3: Extensivierung von Grünlandstandorten – Direkte versus indirekte Auswirkungen auf Bodenmikroorganismen (Multi-Grasland-Landnutzungsversuche)
Die Extensivierung der Landnutzung kann Veränderungen in mikrobiellen Gemeinschaften und in den von ihnen regulierten Bodenfunktionen bewirken. Die neu eingerichteten Multi-Grasland-Landnutzungsexperimente (Abb. 3) werden es ermöglichen, die direkten und indirekten Mechanismen zu identifizieren, die zu Veränderungen der mikrobiellen Abundanz und Funktion beitragen können.