Treibende Prozesse von Rekrutierung und Diversität in Symbiosen: Wie verändern sich Pilz-, Algen- und Bakterien-Gemeinschaften des Flechten-Holobionten entlang eines Landnutzungsgradienten?
Wälder beherbergen vielfältige mikrobielle Gesellschaften aus Algen, Pilzen und Bakterien in einer Vielzahl von Mikrohabitaten. Vertreter dieser mikroorganismischen Gruppen können auf fast jedem Untergrund gefunden werden, wie zum Beispiel Böden und Baumrinde. Flechten sind eines der ältesten bekannten Beispiele für symbiotische Organismen und werden traditionell als mutualistische Symbiose zwischen einem Pilz und einer Grünalge oder einem Cyanobakterium bezeichnet. In den letzten Jahren haben Wissenschaftler allerdings zusätzliche Mikroorganismen (Pilze, Algen und Bakterien) in Flechten gefunden, wodurch diese Klassifizierung zunehmend infrage gestellt wird und Flechten als komplexe ökologische Einheiten (Holobionten) eingeordnet werden. Diese „Miniatur-Ökosysteme“ geben einen wertvollen Einblick in die Zusammensetzung von Gemeinschaften, Interaktion von Arten und die Koordination von Umweltveränderungen auf der Mikroebene. Bisher wissen wir nur wenig über Interaktionen und Beziehungen zwischen den mikrobiellen Gesellschaften von verschiedenen, aber räumlich nahen Subtraten, wie in Flechten-Holobionten, Biofilmen auf Baumrinde und in Böden. Eine umfassende Untersuchung der Biodiversität ist der erste essentielle Schritt, um testen zu können wie Unterschiede in Wäldern und deren Nutzung sich auf die Zusammensetzungsprozesse und Interaktionen dieser mikrobiellen Gruppen auswirken.
Wir wollen verstehen welchen Einfluss Waldstruktur und -nutzungsintensität auf die Diversität und Struktur mikrobieller Gesellschaften (Pilze, Algen, Bakterien) in Wäldern haben. Deshalb werden wir die mikrobiellen Gesellschaften auf Baumrinde, in baumbewohnenden Flechten und in Böden untersuchen. Unser Projekt wird Einblicke in die Häufigkeit und Stabilität der biotischen Interaktionen zwischen verschiedenen mikrobiellen Gesellschaften entlang von Umweltgradienten, in Rekrutierungsprozesse von Mikroorganismen in den Flechten-Holobionten und in Beziehungen zwischen Gesellschaften über und unter der Erdoberfläche liefern.
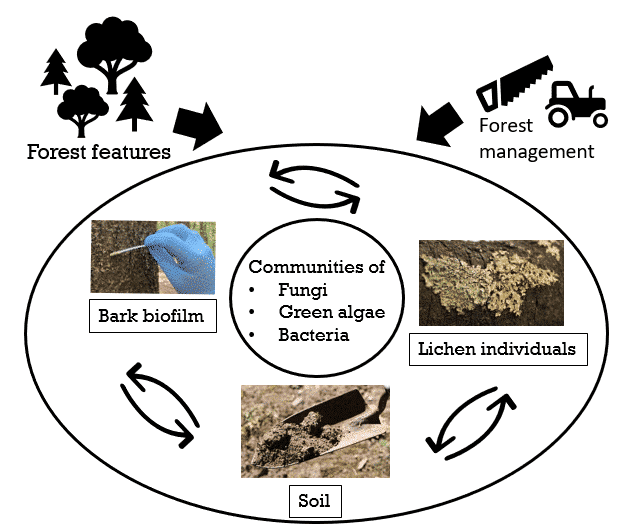
- Sammlung von Biofilmproben von Baumrinde und Bodenproben (Kooperation mit Kernprojekten Boden und Mikroorganismen) auf allen 150 Wald-EPs. Sammlung epiphytischer Flechten von ausgewählten Wald-Plots.
- DNA-Isolation und PCR von Pilzen (ITS), Algen (ITS) und Bakterien (16S).
- High-Throughput Metabarcoding (Illumina MiSeq) zur Untersuchung mikrobieller Diversität.
- Untersuchung der Artenvielfalt und potenzieller Gemeinsamkeiten zwischen Holobiont und Biofilm
- Modellierung von Änderungen der Diversität und biotischer Interaktionen entlang eines Waldnutzungsgradienten.